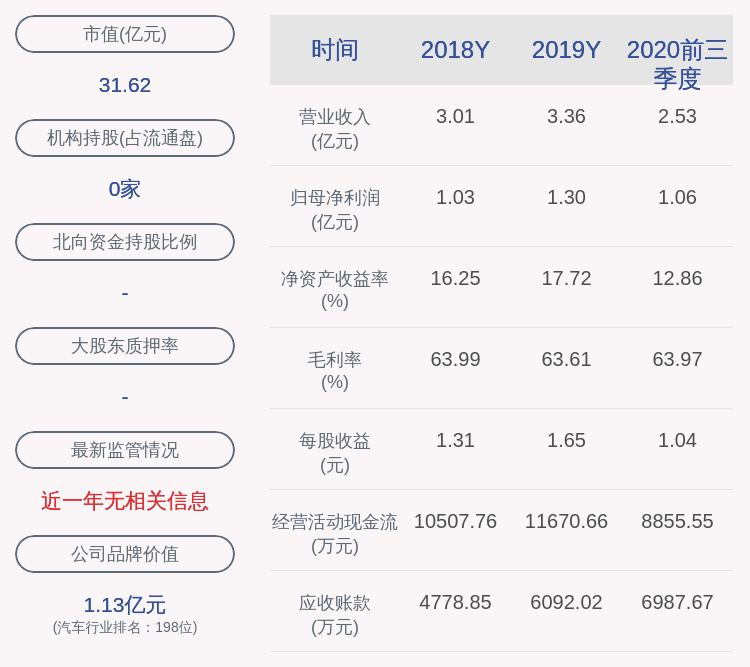
埃尔法哥哥Bionumerics软件的多位点VNTR分析
多位点VNTR分析
多位点VNTR分析(MLVA)是一种基于串联重复序列可变拷贝数(VNTR)对微生物分离株进行亚型分型的分子分型方法 。 即使在高度相关的细菌菌株中 , VNTR通常表现出大拷贝数目的不同 。 对于一组选定的串联重复序列 , 拷贝数分析揭示了微观进化关系 。
本文插图
在实践中 , 选择的VNTR位点对于所研究的生物具有足够的互补性 , 并且在每个VNTR的串联重复序列外设计保守引物 。 因此 , 每个PCR-扩增子的碱基对的大小是串联重复序列的大小加上两端的偏移的总和 。
本文插图
出于经济原因 , 有时会合并多个VNTR , 即用相同的染料标记它们并以混合物的形式装入毛细管测序仪的同一列中 。 条件是混合的VNTR PCR产物的大小范围不重叠 。 例如 , 使用4种染料和2种不重叠的VNTR , 每个毛细管电泳可以确定6种VNTR(一种染料包含用于大小计算的参考marker) 。
本文插图
Bionumerics中MLVA分析
BioNumerics软件提供了用于多位点VNTR分析的全自动工作流程 , 该流程从原始毛细测序仪图谱文件或预处理峰表(Applied Biosystems和Beckman)开始 。 最初必须在数据库中输入MLVA设置 。 这涉及进入样品池的规则:样品池是在同一毛细管中一起加载的VNTR扩增产物的混合物 。这包括使用的不同染料以及(可选)具有不重叠大小范围的兼容VNTR 。 因此 , 每个VNTR由池、染料和(可选)大小范围定义 。 大小范围由重复长度、偏移量和复制范围定义 。 因此 , 该软件确切知道应在哪个大小范围内查找特定的VNTR 。 请注意 , 复制范围仅在不同的VNTR与同一染料合并的情况下才是必需的 。
本文插图
本文插图
如果是原始色谱图文件(AB , Beckman) , 则该软件可以使用用户定义的解析字符串从文件名自动解析池、染料和菌株信息 。
对于GeneMapper或Beckman峰表文件 , 将从制表符分隔的峰表中自动解析此信息(请参见下面的示例) 。
本文插图
稳健而可靠的方法 , 与仪器类型无关
由于仪器、染料和毛细管柱的不同 , 根据拷贝数测得的VNTR扩增子大小通常与理论大小有所不同 。 因此 , 可以以bp为单位输入容忍度 。 显然 , 容忍度应始终小于RepeatSize / 2 。 如果重复量较小 , 则在不同仪器类型之间进行比较时 , 计算出的拷贝数可能会在系统上不同 。 为了解决这个问题 , BioNumerics提供了创建VNTR映射的方法 , 即从观察到的大小到每个VNTR和每个VNTR拷贝的实际大小的映射 。 这种VNTR可确保不同仪器、操作规程、染料、色谱柱等之间的兼容性 。
全自动的拷贝数分析
输入VNTR和解析规则的设置后 , 该软件可以自动处理数千次MLVA运行 , 从而创建报告 , 列出未分析的VNTR , 发现多个峰以及任何其他问题 。 报告可以将与期望值的偏差显示为绿色到红色(下面 , 上面的图像)、左右偏差(蓝色和红色 , 分别是(中间的图像)或仅错误和警告(下面的图像) 。
本文插图
推荐阅读
- 「」儿童节就送阿尔法蛋学习手表,安全贴心又能学习
- -悟空哥哥-618年中大促力来袭,iQOO Neo3应该是你不容错过的5G手机
- 埃尔法哥哥面对用户需求与AI技术之间的不平衡,AI产品经理该如何做?
- 埃尔法哥哥谁说机器学习难?它在这朵云上就没有门槛
- 埃尔法哥哥Python基础语法之“数据应用”
- 埃尔法哥哥一个例子就能读懂大数据,原来数据分析能在这些行业里使用
- 埃尔法哥哥C++程序员的职业生涯规划
- 埃尔法哥哥MAML-Tracker:用目标检测思路做目标跟踪?小样本即可得高准确率丨CVPR 2020
- 埃尔法哥哥数据规范之代码表
- 埃尔法哥哥U盘插在手机的充电器上会怎么样?为什么?